Mehrebenenanalyse mit R (HLM mit R)
– Umwandlung vom Wide-Format in's Long-Format
Arndt Regorz, Dipl. Kfm. & MSc. Psychologie, 14.09.2023
Eine Voraussetzung zum Schätzen eines Mehrebenenmodells ist, dass die Daten im long-Format vorliegen. Das heißt, dass es eine Zeile je Beobachtung gibt. Für Querschnittsdaten ist das häufig sowieso gegeben. Längsschnittdaten hingegen werden häufig ursprünglich im wide-Format gespeichert. Das heißt, dass in einer Zeile alle Beobachtungen einer Person stehen.
Dieses Tutorial zeigt einen möglichen Weg, mit R die Daten vom wide-Format in das long-Format umzustrukturieren. Dies ist jedoch nicht der einzige Weg; es gibt noch andere R-Funktionen, mit denen dasselbe erreicht werden kann.
Video
(Hinweis: Mit Anklicken des Videos wird ein Angebot des Anbieters YouTube genutzt.)
Erzeugen Beispiel-Dataframe
Zunächst erzeuge ich einen Beispiel-Dataframe, an dem ich die Funktionsweise demonstrieren werde (dieser Schritt entfällt bei Ihnen natürlich, Sie nehmen direkt Ihre Echtdaten).
fallnr <- c(1,2,3,4,6,7)
alter <- c(43,42,53,24,35,38)
geschlecht <- c(1,0,1,1,1,0)
uv_t1 <- c(4,5,2,5,8,3)
uv_t2 <- c(4,2,5,5,8,4)
uv_t3 <- c(1,4,3,4,8,3)
av_t1 <- c(1,4,9,5,8,5)
av_t2 <- c(4,3,5,2,3,8)
av_t3 <- c(4,2,2,5,6,4)
daten_wide <- data.frame(fallnr, alter, geschlecht, uv_t1, av_t1, uv_t2,
av_t2, uv_t3, av_t3)
daten_wide

Im long-Format wollen wir, dass die Personenvariablen in jeder Zeile wiederholt werden (hier: fallnr, alter, geschlecht). Außerdem sollen in jeder Zeile von den Messwiederholungsdaten nur die Daten eines Zeitpunktes stehen. Also z.B. entweder uv_t1 und av_t1, oder uv_t2 und av_t2, oder uv_t3 und av_t3. Und zusätzlich brauchen wir dann eine Variable für den Messzeitpunkt.
Umwandlung in das long-Format
Zur Umwandlung in das long-Format verwende ich hier die reshape() Funktion, und zwar mit dem einfachsten Aufruf. Man kann bei der Benennung der umzustrukturierenden Variablen mit der Funktion manchmal auch noch etwas automatisieren; wenn Sie darüber mehr wissen wollen, nutzen Sie die Hilfe-Funktion, also z.B. ?reshape().
Hier ist die eigentliche Umwandlung der Daten:
daten_long <- reshape(daten_wide, idvar = "fallnr", direction ="long",
varying=list(c("uv_t1", "uv_t2", "uv_t3"),
c("av_t1", "av_t2", "av_t3")),
v.names =c("uv", "av"))
Die hier verwendeten Parameter der reshape() Funktion haben dabei folgende Bedeutung:
- Zunächst wird der Name des Dataframes übergeben, der umstrukturiert werden soll (daten_wide).
- idvar gibt die Variable an, in der die Level-2 Einheiten kodiert sind. In einer Längsschnittstudie sind das in der Regel die Nummern der Versuchspersonen, hier "fallnr" (Anführungszeichen nicht vergessen!)
- direction gibt die Umwandlungsrichtung an, hier von wide nach long, also "long".
- varying gibt an, welche Variablen/Spalten sich zwischen den Zeitpunkten unterscheiden. Wenn man mehrere Variablen hat, dann gibt man hier die Namen als Liste von Vektoren an.
Wenn man weitere Messzeitpunkte hätte, dann würde man die einfach ergänzen, z.B. c("uv_t1", "uv_t2", "uv_t3", "uv_t4") bei 4 Messzeitpunkten für die UV, analog für die AV.
Wenn man weitere Variablen hätte, dann würde man eine weitere Zeile innerhalb der list() Funktion ergänzen, z.B. c("med_t1", "med_t2", "med_t3") bei einem zusätzlichen Mediator. - v.names gibt an, wie die Spalten heißen sollen, in die dann die messwiederholten Daten geschrieben werden sollten, und zwar in der gleichen Reihenfolge, wie sie unter varying aufgeführt worden sind.
Bei weiteren Variablen würde einfach ein weiteres Element im Vektor ergänzt werden, z.B. im obigen Beispiel mit der zusätzlichen Mediatorvariable c("uv", "av", "med").
Wenn wir uns das Ergebnis ansehen, haben wir die gleichen Daten im long-Format.
head(daten_long, 8)
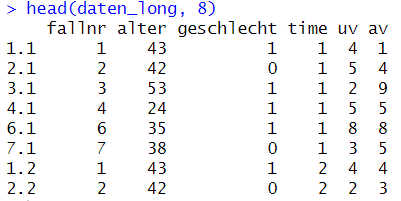
Damit könnten wir jetzt eine Mehrebenenanalyse durchführen. Optisch finde ich aber noch unschön, dass nicht alle Beobachtungen einer Person untereinander stehen und dass die Variable zum Messzeitpunkt (time) soweit rechts steht. Das könnte man noch anpassen.
library(tidyverse)
daten_long_format <- daten_long %>%
arrange(fallnr)%>%
select(fallnr, time, everything())
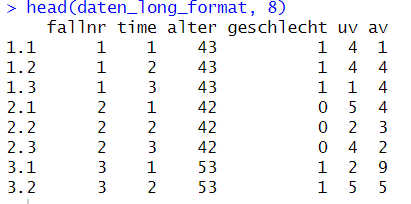
head(daten_long_format, 8)
Hier das m.E. übersichtlichere Ergebnis: